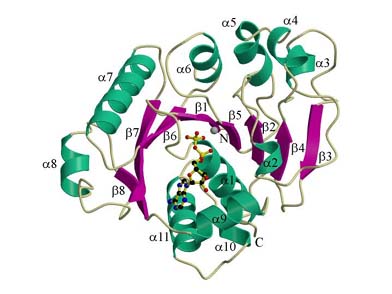
 図はこうして決めたMinDの構造を模式的に
表したものです.緑のリボンがαヘリックスという螺旋構造を,紫のリボンがβ
シートというシート構造を示しています(図をクリックすると活性部位の詳細な構造
が見られます).
図はこうして決めたMinDの構造を模式的に
表したものです.緑のリボンがαヘリックスという螺旋構造を,紫のリボンがβ
シートというシート構造を示しています(図をクリックすると活性部位の詳細な構造
が見られます). 古細菌は進化系統樹上で,大腸菌などの属する真正細菌と真核生物の中間に 位置すると考えられています(例えば総合研究大学院大学の 原生生物情報サーバー 参照).
近年のゲノム塩基配列解析の進行に伴い,古細菌の有する様々な蛋白質群が 真正細菌と真核生物の混合型であることが明らとなり,古細菌の蛋白質の構造を 解明することの重要性が一段と認識されるようになってきています.そうした状 況下,本研究ではすべての生物が共通して有する特徴である細胞周期に着目し, X線結晶構造解析法によって古細菌 Pyrococcus horikoshii OT3 の細胞分 裂に関わる蛋白質群の一つであるMinDという蛋白質の結晶構造を ADP-Mg2+複合体 として分解能2.3Åで解析しました.
 図はこうして決めたMinDの構造を模式的に
表したものです.緑のリボンがαヘリックスという螺旋構造を,紫のリボンがβ
シートというシート構造を示しています(図をクリックすると活性部位の詳細な構造
が見られます).
図はこうして決めたMinDの構造を模式的に
表したものです.緑のリボンがαヘリックスという螺旋構造を,紫のリボンがβ
シートというシート構造を示しています(図をクリックすると活性部位の詳細な構造
が見られます).
バクテリアには細胞内に潜在的な細胞分裂部位が3ヶ所存在しています.これ らのうちの2ヶ所は細胞の両極近傍に存在し,残る1ヶ所の分裂部位は細胞中央に 存在します.両側の2ケ所で細胞分裂が起こると核を有しない細胞(minicell) が生じてしまいます.ですから,バクテリアは細胞のまん中にある正常な分裂部 位で細胞分裂が行われるための調節機構を有しています.
正常な細胞分裂は核様体(nucleoide)が分裂した後にこの分裂部位に細胞 分裂蛋白質FtsZ がリング(Zリング)を形成することで開始されます.大 腸菌 ではminオペロン上にあるMinC,MinD,MinEがこの調節機構を構成しています.
細胞分裂初期にMinE の存在下でMinDはMinCと複合体を形成し,細胞の極から 極へ周期的な振動を行います.これによって細胞両極近傍の2ケ所の分裂部位で のZリングの形成を阻害していると考えられています.また,この際にMinDは振 動運動に必要なエネルギーをATPase活性により生み出しているモーター蛋白質と も考えられています.
MinDのX線結晶構造解析は,遺伝子工学の技術を利用してMinDのアミノ酸のう ちのメチオニン残基をセレノメチオニン化し,シンクロトロン放射光施設 SPring-8 でセレンのX線吸 収端付近の波長のX線を用いて測定し,多波長異常分散法(MAD法) で解析しました.