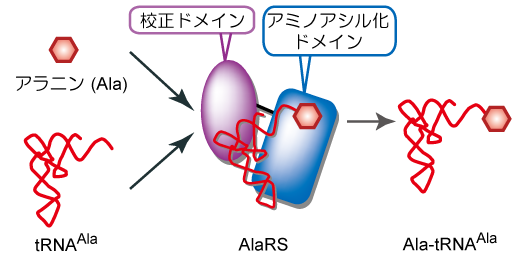
Fig. 1 AlaRSのアミノアシル化反応モデル
タンパク質合成は,リボソーム上でmRNAのコドンと相補的なアンチコドンを持つtRNAを介して,アミノ酸に翻訳されることにより行われます.tRNA構造の一端にはアンチコドンループがあり,mRNAのコドンに特異的に結合します.もう一端にはアクセプターステムがあり,この3'末端にアミノ酸が結合し,アミノアシルtRNA (aa-tRNA) になります.リボソーム上に存在するmRNA内のコドンに対応したアンチコドンを持つaa-tRNAが結合することで,mRNAの遺伝情報を正確にアミノ酸に置換したポリペプチド鎖が作られ,タンパク質合成が行われます.
アミノアシルtRNA合成酵素 (aaRS) は,タンパク質合成において,アミノ酸とそのアミノ酸に対応したアンチコドンを持つtRNAを結合させる,アミノアシルtRNAの生成反応を触媒する酵素です.aaRSの一種であるアラニルtRNA合成酵素 (AlaRS) は,アミノアシル化ドメインにてアラニン (Ala) と,Alaに対応したアンチコドンを持つ tRNA (tRNAAla) を特異的に結合させ,Ala-tRNAAla を合成する機能を有しています (Fig. 1) .

Fig. 1 AlaRSのアミノアシル化反応モデル
タンパク質合成を正確に行うには,AlaRSがAlaとtRNAAlaを正確に結合させることが必要不可欠ですが,アミノアシル化ドメインはある確率でAlaに構造が似ているグリシン (Gly) やセリン (Ser) を誤ってtRNAAlaに結合させ,Gly-tRNAAla やSer-tRNAAlaを生産してしまうことがあります.これらのアミノアシルtRNAがタンパク質合成に利用されると,タンパク質に変異が導入され様々な疾患の原因となり,生命活動に支障をきたしてしまいます.
そこでAlaRSは,アミノアシル化ドメインに加えて校正ドメインを保持しており,Gly-tRNAAla やSer-tRNAAla のアミノ酸を加水分解によって速やかに除去する校正機構を有しています (Fig. 2) .これにより,AlaRSは2段階のアミノ酸選別機構によって目的のAla-tRNAAla のみを高い精度で合成しています.
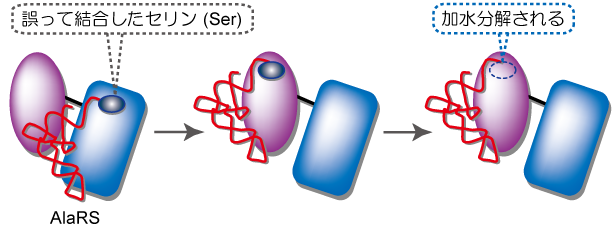
Fig. 2 AlaRSの校正反応モデル
これまでにAlaRSの構造解析に関しては,アミノアシル化ドメインのみの構造や,AlaRS校正ドメインのホモログであるAlaXの構造など部分的な構造は明らかにされていましたが,AlaRSの全体構造は解明されておらず,ドメイン間の相対配置やtRNAとの結合様式など,未解明な課題が多く残っていました.そこで我々のグループでは,超高度好熱古細菌Pyrococcus horikoshii由来のC末端の二量体化ドメインのみを削除したAlaRS (AlaRSΔC) のX線結晶構造解析および生化学実験を行い,実際に各ドメインが働く際の分子機能を明らかにすることを目的としました.
しかし,AlaRSは扱いが困難なタンパク質であり,様々な結晶化条件検討を行いましたが,良質な結晶は得られず,AlaRSΔCの構造決定は非常に困難でした.そこで,AlaRSΔCのリジン残基に還元的メチル化修飾を施した結果,抜本的な結晶性の改善に成功したので,つくばにある高エネルギー加速器研究機構のPhoton Factoryおよび西播磨にあるSPring-8にてX線回折実験を行い,2.16 ÅのX線回折データを得て,AlaRSΔCの高分解能構造の決定に成功しました (Fig. 3) .
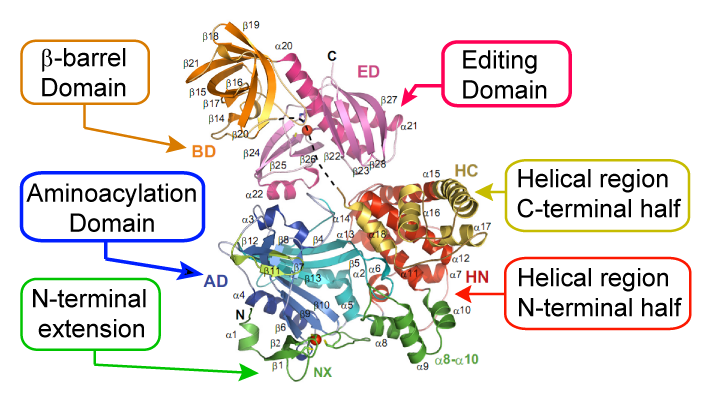
Fig. 3 Pyrococcus horikoshii由来AlaRSΔCの全体構造 (PDB ID: 2zze)
得られたAlaRS全体構造中では,NX内にシステイン4 残基で構成される亜鉛結合モチーフ (β1,β2間のCys-20,23,ロングループ内のCys-37,42) が存在し,結合モチーフ内に亜鉛イオンが存在することが明らかになりました (Fig. 4) .このNX亜鉛結合モチーフは,AlaRSのN末端構造が安定化するのに寄与していると予想されます.
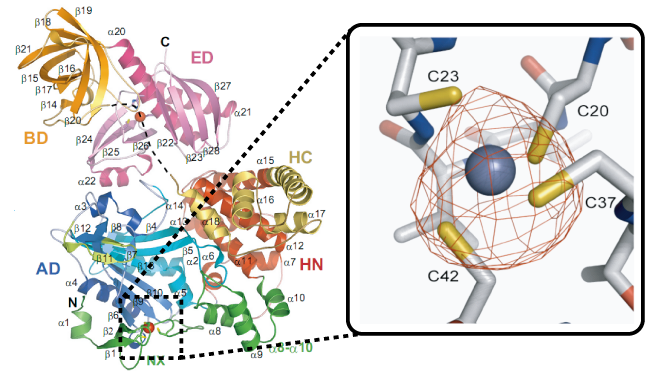
Fig. 4 NXドメインのCys4亜鉛結合モチーフ
青い球体は亜鉛イオンを表している.表示されている異常分散差フーリエマップは5σ以上を表している.
次に,アミノアシル化ドメイン (AD) において,AlaRSの生成物であるAla-AMPのアナログである5'-O-(N-l-alanyl-sulfamoyl)-adenosine (AlaSA) が結合した複合体構造解析を行い,AlaSAがアミノアシル化活性部位に結合している様式を明らかにしました (Fig. 5) .構造によると,Alaのアミノ基をD248が,リン酸基の酸素原子をR128とG250が,リボースのヒドロキシ基をE210が,プリン塩基をD131とF142が認識していることが明らかになりました.また,T213が約120度回転してAlaSAのリン酸基を認識していることが明らかになりました.
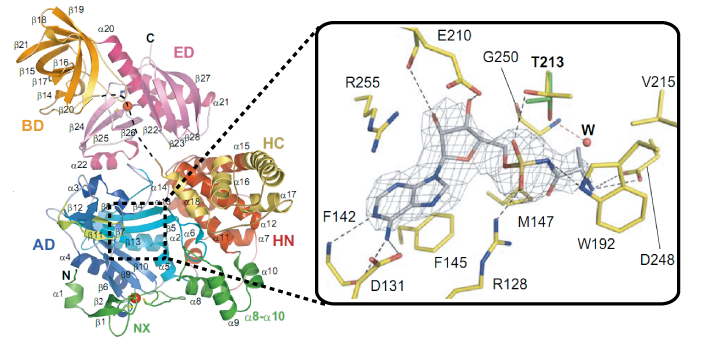
Fig. 5 AD内のアミノアシル化ドメイン活性部位とAlaSA (PDB ID: 2zzg)
黄色はAlaRSΔC,銀色はリガンドのAlaSA,赤い球体のWは水分子を表している.表示されている電子密度図は分解能3.1 Å で,3σ以上を示している.
さらに,校正ドメイン (ED) の活性部位において,亜鉛結合モチーフ (613His XXX His617,717Cys XXX His721) にて亜鉛イオンが配位していることが明らかになりました (Fig. 6) .本研究室の過去の研究により,AlaRS校正ドメインホモログであるAlaX-Sにおいては,活性部位内の亜鉛イオンが校正活性に不可欠であることが明らかになっています.一方,AlaRSのEDと高い相同性をもつスレオニルtRNA合成酵素 (ThrRS) においては,校正活性部位にAlaRSと同様の亜鉛結合モチーフは存在していますが,亜鉛イオンは活性に必要でないことが報告されています.AlaRSにおいて亜鉛イオンが校正反応に必要であるか確かめるために,3H標識したSer-tRNAAlaを用いた校正活性測定を行いました (Fig. 7) .その結果,AlaRS ED活性部位内の亜鉛イオンは,校正反応を促進するものの,不可欠ではないことが明らかになりました.
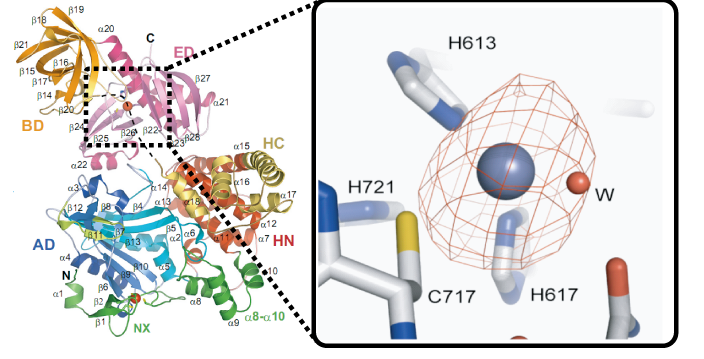
Fig. 6 校正ドメインの亜鉛結合モチーフ
青い球体は亜鉛イオン,赤い球体は水分子を表している.表示されている異常分散差フーリエマップは5σ以上を表している.
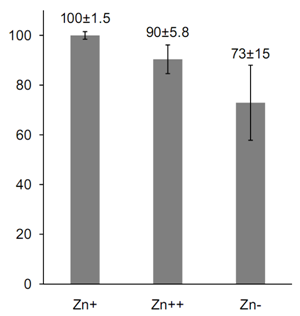
Fig. 7 AlaRSΔCの校正活性測定
Zn+は亜鉛イオンを結合させたAlaRSΔC,Zn++は10 μM亜鉛イオンを加えたAlaRSΔC,Zn-は10 mM EDTA を加え,亜鉛イオンを取り除いたAlaRSΔCを表している.