Ala-tRNAAla校正過程におけるアラニン識別の分子メカニズム
1.遺伝暗号の成り立ち
タンパク質は生物を構成する主要な分子であり,ほとんどの生命活動において中心的な役割を果たす最も重要な分子のひとつです.タンパク質は20種のアミノ酸が順番につながった構造をしており,さらに3次元的に複雑に折れたたまることでさまざまな固有の機能を発揮しますが,このアミノ酸の配列が不適切であった場合,正しく機能を発揮することができません.タンパク質のアミノ酸配列情報は遺伝暗号と呼ばれる規則に基づいて遺伝子上にDNAの3つの塩基の組(コドン)の配列として保存されています.蛋白質が合成される際には,DNAの塩基配列がまずmRNAと呼ばれるDNAと非常によく似た分子に写し取られた(転写)後に,mRNAがリボソームと呼ばれる巨大なタンパク質合成機械に運ばれて,アミノ酸の鎖へと変換(翻訳)されます(図1).この過程において重要な役割を果たすのがアミノアシルtRNA(aa-tRNA)という分子です.aa-tRNAは一方の末端に3つの塩基の組(アンチコドン),他方の端にはアンチコドンに対応するアミノ酸を有しているため,アンチコドンがmRNA上のコドンと相補的な塩基対を形成することでコドンをアミノ酸へと変換するアダブター分子として働きます.言い換えるとaa-tRNAは遺伝暗号のルールそのものを担っている分子であり,tRNAのアンチコドンと付加されるアミノ酸が厳密に正しく対応していないと目的のタンパク質が正確に合成されずに,生命活動に支障をきたしてしまいます.
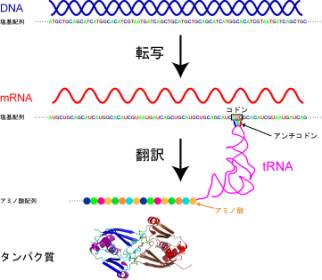
図1:遺伝情報からタンパク質が合成される過程の略図.mRNA上のコドンはtRNAを介してアミノ酸へと変換される.
2.aa-tRNA合成酵素(aaRS)の校正機構
aa-tRNAはそれぞれのアミノ酸に対応する20種のaa-tRNA合成酵素(aaRS)と呼ばれるタンパク質によってアミノ酸がtRNAに付加される事によって合成されます.遺伝暗号を正確に維持するために,aaRSは40種余りのtRNAと20種のアミノ酸の中から目的の数種のtRNAとただひとつのアミノ酸を厳密に識別しなくてはなりません.tRNAに関しては,tRNAが大きな分子であり広い接触面積を確保できることから,分子内のさまざまな場所にあるアイデンティティ決定因子と呼ばれるそれぞれのtRNAに特徴的な構造を認識することで目的のものを比較的容易に識別することができますが,アミノ酸は非常に小さな分子であり互いによく似通ったものが多いことから厳密に識別するのは容易なことではありません(図2A).aaRS自身がアミノ酸で構成されるタンパク質であることを考えると困難さが伺えるかと思います.aaRSのアミノアシル化サイトは目的以外のアミノ酸のほとんどを十分な精度で除外することができますが,目的のアミノ酸によく似たアミノ酸を完全に除くことはできず,そのエラー率は最大で1/200余りにもなります.これは単純に考えると,一般的なタンパク質が200〜300程度のアミノ酸からなっていることことから,タンパク質1分子を合成する際に必ず1つ程度の翻訳エラーが生じることを表しています.実際の細胞内の翻訳エラー率は<1/3000であるといわれており,aaRSの精度はこれを下回っている必要があります.そのため一部のaaRSはアミノアシル化サイトのすぐ近くに校正サイトと呼ばれる別の活性部位を有しており,間違って付加されたアミノ酸のみを特異的に認識して速やかに加水分解する機構を有しています(図2B).このような2段階のアミノ酸選別機構をaaRSの校正機構といい,これによってaaRSは非常に高い精度を維持しています.校正機構を有するほとんどのaaRSはアミノアシル化部位で目的のアミノ酸より大きなアミノ酸を主として立体障害により排除し,校正サイトでは誤って付加された目的のものより小さなアミノ酸を認識・加水分解する,いわゆる「2重ふるい説」に則って目的のaa-tRNAのみを高い精度で合成します.
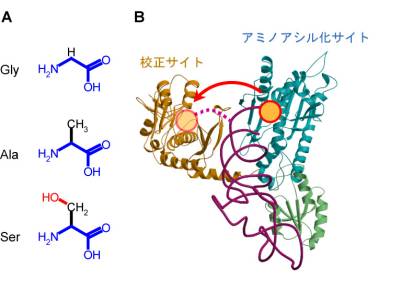
図2:(A)アミノ酸の構造.上からグリシン,アラニン,セリン.共通した構造(主鎖)を青で示した.(B) aaRSの校正機構のイメージ.アミノアシル化サイトで誤って取込まれたアミノ酸(黄色い○)は速やかに隣の校正サイトで認識・加水分解される.
3.アラニル-tRNA合成酵素(AlaRS)の校正機構
アラニル-tRNA合成酵素(AlaRS)はその名のとおりアラニン(Ala)のアンチコドンを持つtRNA(tRNAAla)にAlaを付加するaaRSのひとつです.AlaRSのアミノアシル化サイトはAlaの他によく似たセリン(Ser)やグリシン(Gly)を誤って取込んでしまいますが,校正サイトにおいて誤った産物のみを特異的に加水分解することで精度を維持しています.しかし,Glyは確かにAlaより小さな分子であり2重ふるい説によく当てはまりますが,Serに関してはAlaよりも大きく,しかも親水性のヒドロキシル(OH)基を有していることから従来の説ではうまく説明することができません(図2左).
このようなメカニズムを知るためにはAlaRSの立体構造を解析し,アミノ酸の識別機構を視覚的に調べるのが最も有効な手段ですが,AlaRSは比較的扱いが困難なタンパク質であり,なかなか容易ではありません.そこで我々の研究グループはAlaRS校正ドメインの断片遺伝子であるAlaXに着目し(図3),このAlaXとSerの複合体のX線結晶構造解析を行うことでAla-tRNAAlaの校正機構を詳細に明らかにすることに成功しました.AlaXのような断片的な遺伝子はaaRSが進化の過程で校正機構を獲得してきた名残であると考えられ,実際にこのような遺伝子がさまざまな生物のゲノムに数多く見出されてきています.
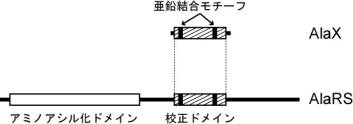
図3:AlaRS,AlaXのドメイン構造.AlaXは校正ドメインに相当する部分のみを持つ.
4.AlaX-Ser複合体の構造
X線回折データは、つくばにある高エネルギー加速器研究機構のPhoton
Factoryおよび姫路にあるSPring-8において収集されました。AlaXの全体構造は図に示すように非常にコンパクトなおにぎり型の構造をしており,分子のほぼ中央にSerが結合していました.校正サイトは亜鉛結合モチーフとその反対側の親水性ポケットからなっており,Serは主鎖部分を亜鉛結合モチーフ,側鎖部分を親水性ポケットに向けるようにして認識されていました.特に側鎖のg-OH基が水素結合ネットワークにより強く認識しており,この相互作用がSerに対する特異性に重要であると考えられます(図4).
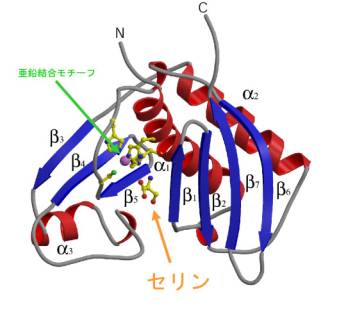
図4:AlaX-Ser複合体の全体構造.
さて,校正サイトではAlaは認識されないわけですが,どのようにしてSerと区別されているのでしょうか?単純に考えるとAlaはSerよりも小さいのでSerが認識される環境ではAlaも認識されるように思われます.この観点で立体構造をよく観察すると,Serのb位の炭素(すなわちAla側鎖が来るべき場所)とThr30側鎖のg-OH基が3.7Åと両者のファンデルワールス半径とほぼ一致する距離にあることが分かります(図5A,B).前者が疎水性,後者が親水性の官能基であることを考えると両者の間には少なからず反発力が生じていることが伺えます.Serはg-OH基が親水性ポケットと非常に強く相互作用しているためこの反発力は問題となりませんが,Alaが校正サイトに結合しようとしても,側鎖b-メチル基とThr30-g-OH基間の反発力によりうまく結合できないことが予想されます.実際,Thr30はAlaX間で非常に高く保存されていることからも,この残基の重要性が伺えます.一方,Glyに関しては側鎖が存在しないため,単純に主鎖部分と亜鉛結合モチーフの相互作用によりやはり校正サイトに結合できるものと考えられます(図5C).このようにAlaX校正サイトでは主に化学的な性質の違いに基づいてアミノ酸を識別している点が,立体障害をベースにしていた従来の2重ふるい説と異なっており,そのためAlaより大きいアミノ酸と小さなアミノ酸を同時に選択的に認識することが可能であると考えられます.
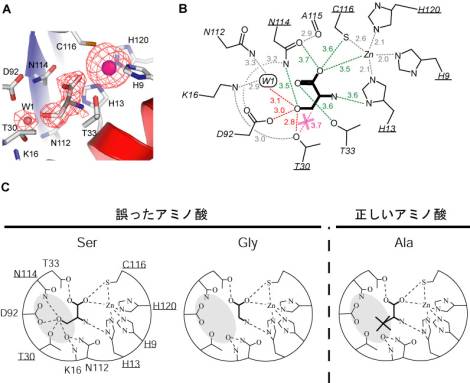
図5:(A)校正サイト付近の構造.赤いメッシュは電子密度を表す.(B)校正サイト付近の模式図.点線の横にある数字は距離(Å)を表す.Serの主鎖部分は主に亜鉛結合モチーフ,側鎖g-OH基は親水性ポケットによって認識されている.Thr30はポケットの「入り口」付近に存在し,b位の炭素と近接している(マゼンタの点線).(C)AlaX校正サイトにおける化学的識別モデル.Serはg-OH基の強い認識,Glyは側鎖がないことにより校正サイトに結合できるが,Alaはb-メチル基とThr30-g-OH基の反発力により排除される(×印).
5.AlaRSにおけるアミノ酸識別機構
さて,構造解析からAlaX-Thr30がAlaとSerの識別に重要であるという化学的識別モデルが考えられるわけですが,では本当にこのモデルが妥当なのか,また,断片的遺伝子であるAlaXで得られた結果がAlaRSにも通用するのか,という疑問がわいてきます.まず1つ目の疑問に答えるために,我々はThr30をバリンに置換したAlaX-T30V変異体を作成し,校正活性に及ぼす影響を調べました.この変異体ではThr30-g-OH基がメチル基に置き換わっているため,ほとんどサイズや形は変わらずに親水基が疎水基に変化しています.我々の化学的識別モデルが妥当であればこの変化によりAla側鎖に対する反発力がなくなるため,校正サイトはSerに加えAlaも認識するようになるはずです.T30V変異体と野生型AlaXのSer-tRNAAla
とAla-tRNAAlaに対する加水分解活性を調べたところ,Ser-tRNAAlaでは両者が同レベルの活性を有していたのに対して,Ala-tRNAAlaに対する活性は変異体でのみ顕著に増加していました(図6A,
B).これはT30V変異導入によりAlaとSerの識別能が失われたことを表しており,Thr30-g-OH基の親水性がAlaとSerの識別に重要であるという我々の説を強く裏付けるものです.次にAlaRS校正サイトでも同様のメカニズムが成立するのか検証するために,AlaX-Thr30に相当する残基であるAlaRS-Gln633を先ほどのT30V変異体と同様にサイズをなるべく変えずに疎水基へと置換したQ633M変異体を作成し,同様の活性測定を行いました.結果,Q633M変異体はT30V変異体とまったく同様にAlaを認識するようになったことから(図6C,D),AlaRSの校正サイトにおいてもやはりAlaXと同様の化学的識別機構によりアミノ酸が識別されているものと考えられます.Gln633もやはりAlaRS間で完全に保存されている残基であり,この残基の重要性を強く裏付けています.
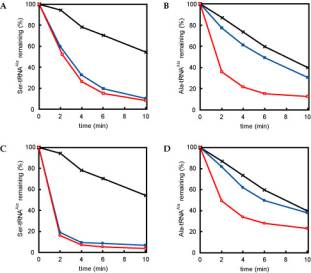
図6:Ser-tRNAAla(A,C),Ala-tRNAAla(B,D)に対するAlaX(A,B)およびAlaRS(C,D)の加水分解曲線.青が野生型,赤が変異体,黒がバックグラウンドを表す.AlaX,AlaRS共にSer-tRNAAlaに対する活性が変化しない(A,C)のに対して,Ala-tRNAAlaに対する活性が変異体でのみ著しく上昇している(B,D).
6.まとめ
aaRSによるアミノ酸の認識はその重要性を考えると,生物の中で行われている反応の中でも最も高い精度が要求される反応のひとつであり,いわゆるDNAワールドの黎明期から現在に至るまで非常に長い時間と淘汰を重ねて極めて高度に洗練されてきた分子認識・識別メカニズムであると考えられます.Alaはその中でも特に小さなアミノ酸であり,炭素ひとつ分程度の非常に小さな違いを驚くべき精度で特異的に識別するメカニズムは,現在の人間の技術では到底まねできるものではありません.今後,更なる研究を続けることでAlaXのような断片的遺伝子の存在意義や,AlaRSとの進化的な関係,あるいはAlaRS自身のさらに詳細なメカニズムが明らかになることで,生命の持つ意味やその起源に関して知見が得られることが期待されます.